全基因组测序(Whole Genome Sequencing, WGS)是利用高通量测序平台对人类不同个体或群体进行全基因组测序,利用生物信息学手段得到全基因组序列,全面挖掘DNA水平的遗传变异,包括较大的结构变异。测序周期短,数据覆盖均一,可用于拷贝数变异和结构变异的检测、融合基因检测、病毒整合位点检测、非编码区突变检测。为筛选疾病的致病及易感基因,研究发病及遗传机制,以及推断种群进化等提供重要信息。
全基因组测序在基因组的编码区和非编码区对致病突变位点/基因进行筛选或预测,与外显子测序相比, 能够发现更多大的结构变异。全基因组测序同样根据疾病的类型及特点进行取样并设计测序及分析策略。
| 测序平台 |
测序模式 |
测序深度 |
| Illumina MiSeq/HiSeq/NovaSeq | PE250/PE150 | 150-200 |
| 分析内容 | ||
| 标准分析 | 个性化分析 | |
|
数据质控 序列比对 变异检测:SNP/InDel/CNV/SV/变异位点筛选 基因组变异Circos图展示 |
多样本变异位点异同分析 致病候选基因筛查预测 新生突变筛选及分析 候选基因富集分析 调控网络分析 肿瘤基因组分析 驱动基因/致病机理分析 易感基因分析 异质性分析 病毒整合分析 个性化图形展示 临床数据整合分析 |
|
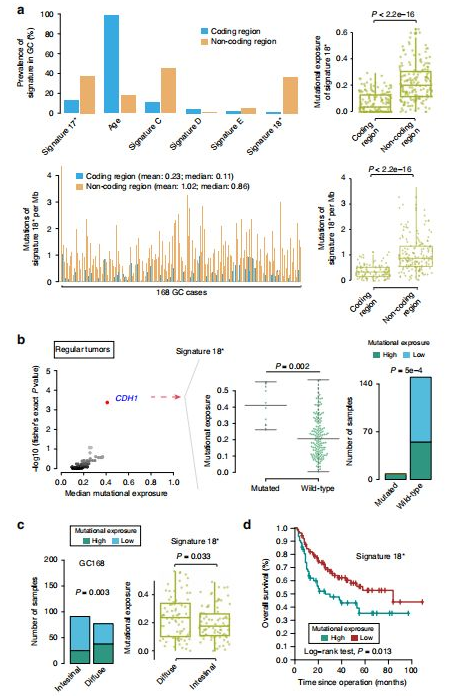
全基因组测序揭示胃癌的串联重复热点和预后突变特征
为了进一步解决胃癌的结构变化和全基因组突变特征,本研究收集来自国际癌症基因组联合会项目的168例未经化疗和放疗的中国胃癌患者,其中弥漫性胃癌77例,肠型胃癌91例。此外,143例肿瘤在胃内的位置明显,并进行全基因组测序,探索中国胃癌人群的SV特征。结果表明在胃癌中发现一些包含CCNE1,EGFR,MYC等驱动基因的复杂SV,还发现了MYC和ZFP36L2超增强子区域的TD热点,且由TD导致的ZFP36L2超增强子的扩增会导致ZFP36L2基因表达上调,高表达的ZFP36L2会促进胃癌细胞的增长,还发现signature18*与预后差有关。