


全基因组关联分析(Genome-wide association study,GWAS)是研究动植物复杂性状的重要手段。通过对自然群体具有丰富遗传多样性的每个个体进行全基因组重测序,结合准确的目标性状的表型数据及统计方法进行全基因组分子标记与性状之间的关联分析,可快速获得影响目标性状表型变异的遗传标记或候选基因。
| 分析内容 | ||
|
下机数据统计 数据质控 高质量数据获取 参考基因组整理 序列比对 Tag数目、分布和测序深度统计 Tag分布的可视化图形 SNP检测 SNP注释 群体分层分析 连锁不平衡距离计算 |
表型整理 表型相关性计算 全基因组关联分析 Manhattan plot QQ plot Peak SNP的统计 候选基因注释 候选基因与已报道GWAS定位结果比较 候选基因与已知QTL比较 候选基因表达量分析 候选基因遗传变异分析 |
|
GWAS解析大豆复杂农艺性状间的调控网络
本研究收集了809份大豆材料并在三个地点对它们进行了两年的表型研究,获得了84个农艺性状。全基因组关联研究鉴定了245个显著的遗传位点,其中95个位点与其他位点存在遗传互作。用显著的遗传位点及其对应的性状构建性状间的遗传调控网络。性状与显著信号之间直接关联,信号与信号直接通过inter-LD关联。结果发现同一类的性状趋于聚在一起,一些显著信号与多个性状相关联,因此探索了这些区域是存在一因多效还是紧密连锁。结果发现是一因多效的作用。
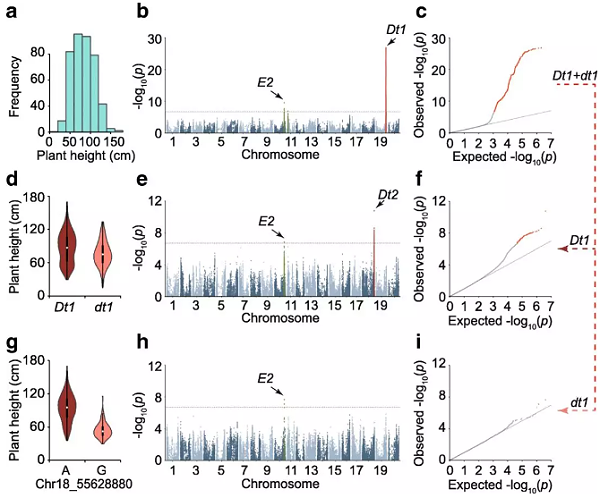
大豆株高的GWAS