


基于PacBio三代测序平台的全长转录组测序(Iso-Seq),可直接获得mRNA的全长序列,可更为准确地解读mRNA的结构信息。通过全长转录组测序,可克服无参考基因组物种转录本拼接短、信息不完整的难题,获取全长mRNA序列,实现同源异构体分析,单碱基变异检测,可变剪接分析,同源基因识别及等位基因识别。
全长转录组实验设计思路可以是单个样品进行测序,与参考基因组进行比较进行结构分析,亦可以对多个样品进行基因结构方面的差异比较分析。
| 分析内容 | ||
|
RoI序列统计 RoI序列长度分布 RoI序列质量分布 RoI Number of Passes分布 转录本分类 聚类与校正 比对及进一步聚类 Known Isoforms注释汇总 Known Isoforms Go注释统计 Known Isoforms KEGG注释统计 Known Isoforms eggNOG注释统计 Novel Isoforms注释汇总 Novel Isoforms NR注释统计 Novel Isoforms GO注释统计 Novel Isoforms KEGG注释统计 |
Discarded Isoforms eggNOG注释统计 Discarded Isoforms注释汇总 Discarded Isoforms NR注释统计 Discarded Isoforms GO注释统计 Discarded Isoforms KEGG注释统计 Discarded Isoforms eggNOG注释统计 融合基因分析 基因结构优化 SNP分析 可变剪切分析 基因表达量统计(结合二代测序数据) 基因表达差异分析(结合二代测序数据) 表达差异基因GO富集分析(结合二代测序数据) 表达差异基因KEGG通路富集分析(结合二代测序数据) 表达差异基因聚类分析(结合二代测序数据) |
|
莲草直胸跳甲全长转录组分析
本研究对空心莲子草进行了全长转录组测序,获得较完整的转录本集合,预测了145个可变剪接事件;27318条简单重复序列,经TransDecoder鉴定获得24,040个ORF,其中有16,205个完整的ORF;预测得到4,198 个lncRNA,为进一步研究莲草直胸跳甲与宿主植物和生态系统之间相互作用的分子机制提供了很好的遗传信息基础。
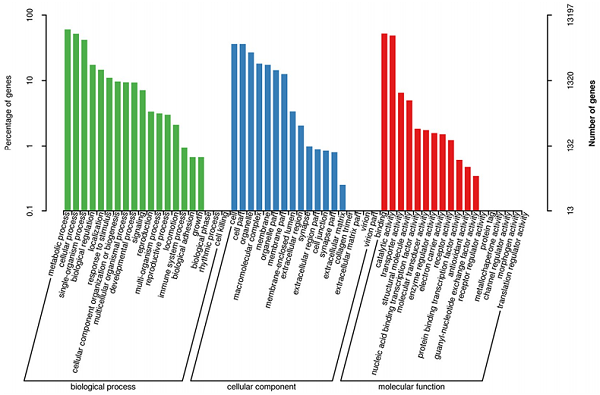
莲草直胸跳甲转录本的GO功能注释